这么多年下来,我确实是没处理过modis影像
这么多年下来,我确实是没处理过modis影像
ytkz读研的时候,研究方法跟modis是不搭边的。工作后也没机会去使用modis。
modis的数据格式是hdf。
hdf文件和nc文件非常像,而我对nc格式的文件算是比较熟悉。
这么多年下来,我确实是没处理过modis。
modis的数据处理是不难的。
昨天看到一个留言说,怎么将modis影像保存为tif格式。
所以,今天的主题就是,讲解怎么将modis影像保存为tif格式。
代码
#!/usr/bin/env python
# -*- coding: utf-8 -*-
# @Time : 2024/10/12 19:22
# @File : hdf2tif.py
'''
modis 数据 由hdf格式转换为tif格式
'''
from osgeo import gdal
import os
import glob
# gdal打开hdf数据集
def modishdf2tif(inpath, outpath = None):
os.chdir(inpath)
file_list = glob.glob("*.hdf")
for i in file_list:
datasets = gdal.Open(i)
# 获取hdf中的子数据集
SubDatasets = datasets.GetSubDatasets()
Metadata = datasets.GetMetadata()
# 打印元数据
for key,value in Metadata.items():
print('{key}:{value}'.format(key = key, value = value))
# 获取要转换的子数据集
data = datasets.GetSubDatasets()[0][0]
Raster_DATA = gdal.Open(data)
DATA_Array = Raster_DATA.ReadAsArray()
print(DATA_Array)
# 保存为tif
if outpath == None:
outpath = inpath
TifName = os.path.join(outpath,os.path.splitext(os.path.basename(i))[0] + '.tif')
geoData = gdal.Warp(TifName, Raster_DATA,
dstSRS = 'EPSG:4326', format = 'GTiff',
resampleAlg = gdal.GRA_Bilinear)
del geoData
if __name__ == '__main__':
in_path = r'D:\modis'
modishdf2tif(in_path)逐行讲解代码
```python
from osgeo import gdal
import os
import glob- 导入GDAL库,用于读取和处理地理空间数据。
- 导入操作系统模块,用于文件系统操作。
- 导入glob模块,用于文件名模式匹配。
def modishdf2tif(inpath, outpath=None):- 定义一个函数
modishdf2tif,接受两个参数:输入路径inpath和输出路径outpath。如果未指定outpath,则默认为None。
os.chdir(inpath)
file_list = glob.glob("*.hdf")- 改变当前工作目录到指定的输入路径。
- 列出当前目录下所有的
.hdf文件,并存储在file_list变量中。
for i in file_list:
datasets = gdal.Open(i)- 对于
file_list中的每一个HDF文件,使用GDAL的Open()方法打开文件。
# 获取hdf中的子数据集
SubDatasets = datasets.GetSubDatasets()
Metadata = datasets.GetMetadata()- 获取HDF文件中的子数据集列表。
- 获取HDF文件的元数据。
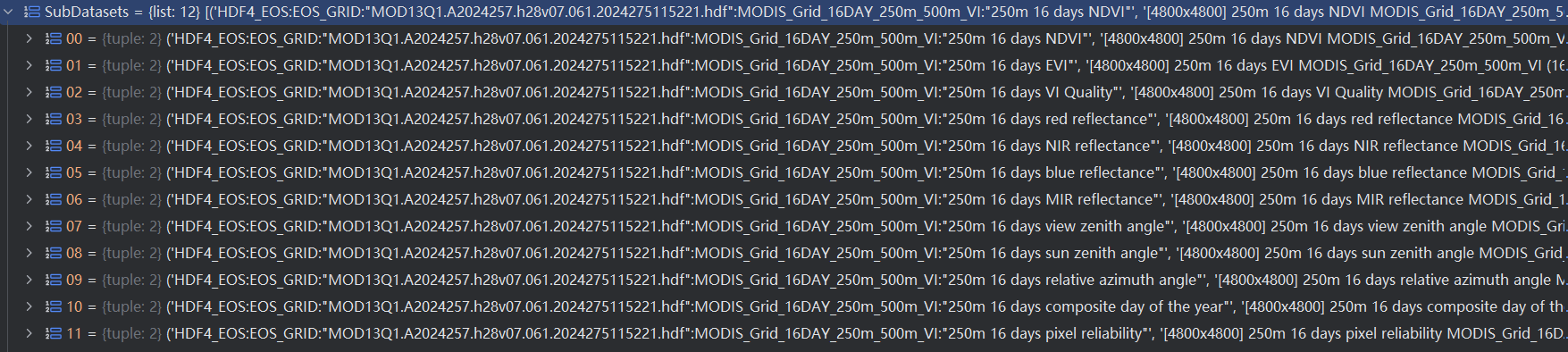
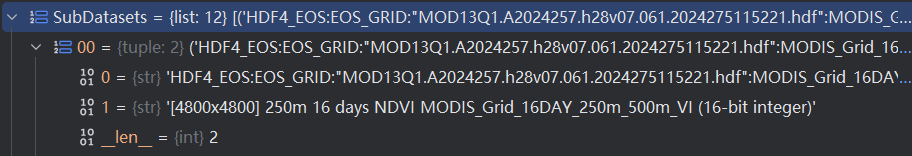
这里可以打个断点,仔细看看子数据集有什么内容。
我截了个图,以这个modis文件为例,SubDatasets存在12个元组元素,如下:
这12个元组,包含了两个字符串,如下:
# 打印元数据
for key, value in Metadata.items():
print(f'{key}: {value}')- 使用
for循环打印HDF文件的所有元数据项。
# 获取要转换的子数据集
data = datasets.GetSubDatasets()[0][0]
Raster_DATA = gdal.Open(data)
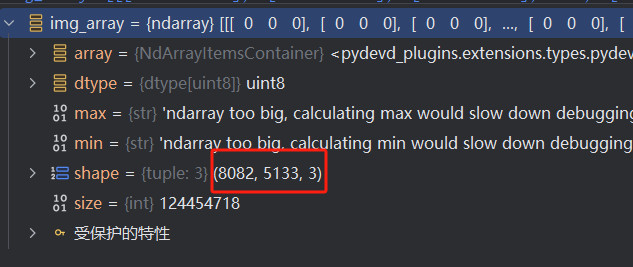
DATA_Array = Raster_DATA.ReadAsArray()
print(DATA_Array)- 选择第一个子数据集的路径。
- 使用GDAL打开这个子数据集。
- 读取子数据集的数据到一个NumPy数组中,并打印这个数组。
# 保存为tif
if outpath is None:
outpath = inpath- 如果没有指定输出路径,则使用输入路径作为输出路径。
TifName = os.path.join(outpath, os.path.splitext(os.path.basename(i))[0] + '.tif')- 使用
os.path.join()构建输出TIFF文件的完整路径。这里先获取HDF文件的基础文件名(不带扩展名),然后加上.tif扩展名。
geoData = gdal.Warp(TifName, Raster_DATA,
dstSRS='EPSG:4326', format='GTiff',
resampleAlg=gdal.GRA_Bilinear)- 使用
gdal.Warp()函数将子数据集重投影并保存为GeoTIFF格式。指定输出坐标系为WGS84(EPSG:4326),格式为GeoTIFF,重采样算法为双线性插值。
del geoData- 删除
geoData对象,释放资源。
if __name__ == '__main__':
in_path = r'D:\modis'
modishdf2tif(in_path)- 当脚本直接运行时,设置输入路径为
D:\modis,并调用modishdf2tif函数。
测试
以一景modis数据为例子,进行代码测试。
输入参数为modis文件所在的文件夹,这样写的目的是方便进行批处理。该modis文件名字为MOD13Q1.A2024257.h28v07.061.2024275115221.hdf
输入文件为一个对应的tif文件。tif文件名字为MOD13Q1.A2024257.h28v07.061.2024275115221.tif
直接使用win自带的图片查看器可以打开这个tif,如下:
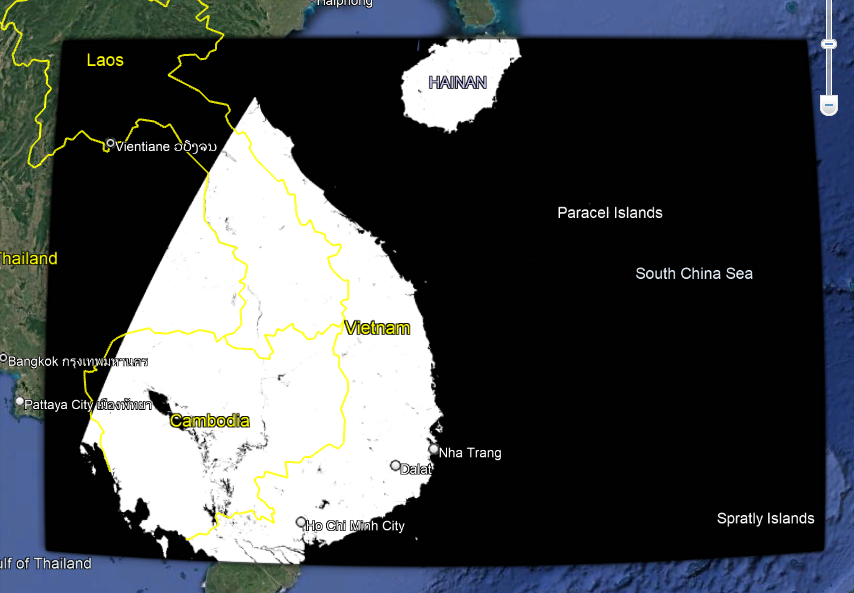
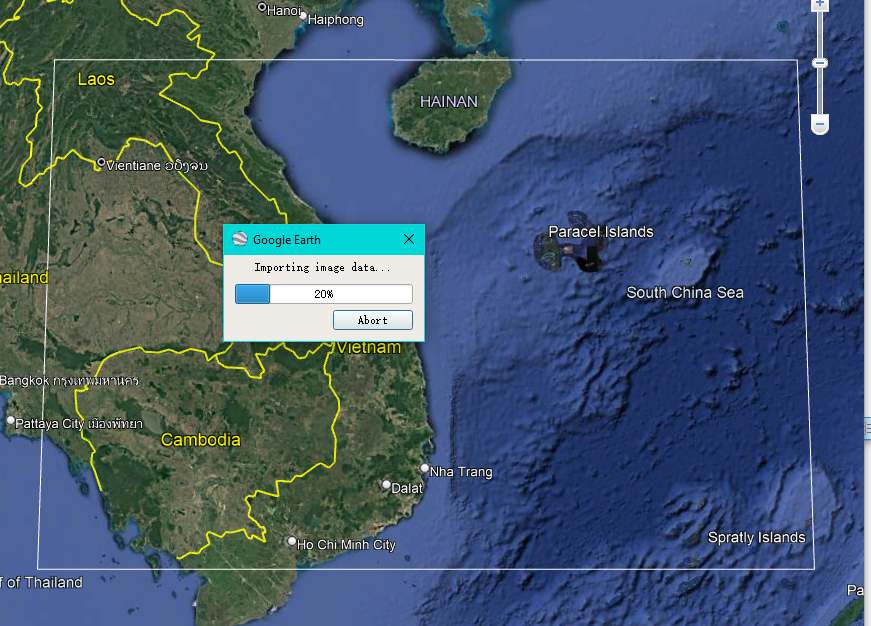
代码运行完毕后,把这个tif文件直接拖拽到google earth,检查数据是否正常(几何定位是否有问题)。
最后把这个tif拖拽到QGIS上,可视化结果如下:
再细说一说
hdf 、nc这两种文件,给我的感觉就是非常相像。而tif相对来说,则是比较简单。这种简单带来的缺点是文件的大小比较大。
这句话怎么理解呢?
比如说,我想把一个100x100的矩阵和一个500x500的矩阵,存放到tif中。
这时有两个做法:
1.将100x100矩阵向上重采样到500x500(文件数据变大)
2.将500x500矩阵向上重采样到100x100(精度损失)
这两种做法都不理想。这时候就凸显出hdf、nc的优越性了。
再比如说,我想把一个100x100的矩阵和一个500x500的矩阵,存放到hdf或nc文件中。
这时,不需要重采样就可以把数据直接存放在hdf\nc文件中。
但是这样的优越性也同样带来另一种麻烦,我称之为 不统一性。
比如我读取海洋二号的数据和读取sentinel-3的数据,写的代码是不可以统一的。
虽然这二者都是NC类型的数据,但是!它们内部的数据读取的名字、属性完全不一样。
我需要慢慢调试、或者慢慢看说明文档才能把它们的数据读取为矩阵,存放早内存里。
以海洋二号为例,它会把经纬度、经纬度间隔分别存放NC中,我们需要把这三个数据读取出来,再插值为网格,才能把数据完整地读取出来。经纬度的名字,在海洋二号中可能是LON和LAT;在sentinel-3中可能就变成了小写的lon、lat。
而今天的modis影像,则是没有类似与lon、lat的属性。
只能说hdf\nc文件是百花齐放一团糟。内部接口不统一是它俩的特性。
方便学习
我把代码和文中测试的modis影像上传到云盘
明天会尝试把这个功能集成到rstool中。